|
newsletter finesettimana #12
Buon venerdì,
questa settimana parliamo della misura più precisa mai effettuata per la costante
di struttura fine dell'atomo di idrogeno, del crollo del radioteloscopio di Arecibo,
dell'assassinio del fisico iraniano Fakhrizadeh, di una nuova tavola periodica degli elementi,
di un'analisi del sangue per diagnosticare precocemente diverse forme di tumore
e dell'approvazione del primo vaccino contro COVID-19 nel Regno Unito.
La notizia che approfondiamo oggi è quella che riguarda l'algoritmo AlphaFold2
sviluppato dalla società londinese DeepMind, che ha dimostrato di essere in grado di calcolare
la struttura tridimensionale di un centinaio di proteine a partire dalla loro sequenza
con accuratezza paragonabile a quella sperimentale.
Buona lettura e al prossimo venerdì (per segnalare questa newsletter
agli amici
ecco il link per l'iscrizione)
|
|
|
SEI PEZZI BELLI
|
1 Misurata con una precisione senza precedenti la costante di struttura fine
Si tratta della costante che stabilisce l'intensità dell'interazione
tra particelle dotate di carica elettrica ed è legata alla distanza tra certe
linee spettrali dell'atomo di idrogeno, misurate sperimentalmente per la prima
volta da Michelson e Morley nel 1887.
Oggi un gruppo di ricercatori del Kastler Brossel Laboratory di Parigi
la ha misurata con un margine di errore di 81 parti per miliardo,
una precisione tre volte superiore a quella della migliore misura finora
disponibile.
Il risultato ottenuto è in accordo con quello che si ottiene
dalla misura di una costante legata al momento magnetico dell'elettrone.
Il fatto che le due stime siano compatibili esclude molti dei modelli
che propongono l'esistenza di una nuova interazione fondamentale oltre alle quattro
che già conosciamo, elettromagnetica, debole, forte e gravitazionale
[Quantamagazine]
3 Il radiotelescopo di Arecibo è crollato
Intorno alle otto del mattino del primo dicembre le
900 tonnellate di strumentazione sospese 300 metri al di sopra
del disco parabolico
del radiotelescopio sono crollate sulla piattaforma, distruggendola.
Due dei tre cavi che sostenevano la strumentazione ancorata a tre torri,
si erano danneggiati il 10 agosto e il 6 novembre.
Giudicando i danni irreparabili, la National Science Foundation americana,
responsabile dell'osservatorio di Porto Rico, aveva deciso di demolire il telescopio,
uno dei più grandi al mondo che ha rappresentato una risorsa fondamentale per i radio astronomi negli ultimi
sessanta anni
[Le Monde]
2 L'assassinio del fisico iraniano Mohsen Fakhrizadeh
allontana la possibilità di ristabilire l'accordo sul nucleare iraniano
Mohsen Fakhrizadeh è stato assassinato in un attacco subito fuori Tehran
e le autorità iraniane hanno finora attribuito la responsabilità a Israele.
Fino al 2003 Fakhrizadeh ha diretto AMAD, un programma segreto per lo sviluppo di armi nucleari.
Ma le agenzie di intelligence israeliane hanno reperito nel 2018 dei documenti
che suggeriscono che lo scienziato abbia ripreso le sue attività di ricerca
dal 2011 clandestinamente in un centro di ricerca della Guardia Rivoluzionaria
iraniana. La morte di Fakhrizadeh allontana la possibilità che il patto sul nucleare,
firmato
nel 2015 con gli Stati Uniti di Obama per limitare le operazioni di arricchimento dell'uranio
in cambio di concessioni economiche e successivamente interrotto dall'amministrazione Trump,
possa essere rivitalizzato quando Joe Biden si insedierà alla presidenza degli Stati Uniti
[Science]
4 Una nuova proposta per ordinare gli elementi chimici su una tavola periodica
La tavola periodica può essere il modo più iconico di ordinare gli elementi, ma non è necessariamente il più utile.
Capita infatti che elementi adiacenti abbiano poco in comune dal punto di vista chimico.
Due scienziati dello Skolkovo Institute of Science and Technology di Mosca
hanno proposto un nuovo modo di disporre gli elementi secondo le loro proprietà
all'interno di composti binari.
Utilizzando questo schema, dicono che sarà possibile prevedere quali fra questi composti binari
avranno caratteristiche simili, aiutando a identificare nuovi materiali
[The Conversation UK]
5 Nella primavera del 2021 il National Health Service inizierà a testare un'analisi del sangue in grado di identificare precocemente 50 diverse forme di tumore
Il test si chiama Galleri e potrebbe rivoluzionare la diagnosi precoce in campo oncologico.
Il servizio sanitario britannico lo inizierà a testare nella primavera del prossimo anno
su un campione di 165 000 cittadini tra i 50 e i 79 anni asintomatici. La speranza è che aiuti a
diagnosticare tumori che non danno sintomi fino a quando non sono molto avanzati e dunque
lasciano scarse probabilità di sopravvivenza, come il cancro al pancreas o alle ovaie.
In questa prima fase i pazienti verranno seguiti fino al 2023 e, se
il test si dimostrerà efficace, sarà realizzato uno studio clinico più grande
che coinvolgerà fino a un milione di persone
[The Guardian]
6 Il Regno Unito approva il vaccino contro COVID-19 prodotto
da Pfizer e BioNTech
Si tratta del primo Paese ad autorizzare l'utilizzo di un vaccino contro
COVID-19. Le vaccinazioni dovrebbero cominciare la prossima settimana
per le categorie a rischio. Il farmaco ha ottenuto l'approvazione
per l'utilizzo in condizioni di emergenza dall'agenzia del farmaco britannica,
la Medicines and Healthcare products Regulatory Authority.
Il Regno Unito ha acquistato 40 milioni di dosi del vaccino di Pfizer e BioNTech
che ha mostrato un'efficacia del 95%, secondo le dichiarazioni delle
compagnie farmaceutiche. Lo schema di priorità che sarà seguito per la campagna vaccinale
verrà reso noto nei prossimi giorni dal Joint Committee on Vaccination and Immunisation
[The Guardian]
|
|
|
DEEPMIND CALCOLA LA STRUTTURA DELLE PROTEINE CON ACCURATEZZA SORPRENDENTE
|
.
'L'intelligenza artificiale trionfa nel calcolo della struttura delle proteine'.
'DeepMind ha risolto una sfida della biologia lunga 50 anni'.
'La soluzione di un mistero della biologia'.
'Uno dei più grandi misteri della biologia sostanzialmente risolto da un'intelligenza artificiale'.
Con questi titoli i maggiori quotidiani del mondo hanno riportato la notizia
che un sistema di intelligenza artificiale, sviluppato dalla società londinese DeepMind,
è stata in grado di determinare la struttura tridimensionale di un centinaio di proteine
con un'accuratezza mai raggiunta prima e paragonabile a quella delle tecniche sperimentali
che finora hanno svelato la maggior parte delle strutture proteiche che conosciamo.
La notizia è arrivata dagli organizzatori della competizione CASP (Critical Assessment of techniques for protein Structure Prediction),
che si tiene ogni due anni e che quest'anno è giunta alla sua quattordicesima edizione. Dal 1994 CASP organizza una sfida
tra decine di gruppi di ricerca nel campo della biologia computazionale chiedendogli di prevedere la struttura tridimensionale
di un campione di proteine e complessi proteici a partire dalla sequenza di amminoacidi che li costituiscono.
Quest'anno AlphaFold2, questo il nome dell'intelligenza artificiale sviluppata da DeepMind, ha vinto di misura
su tutti gli altri partecipanti.
La struttura tridimensionale delle proteine stabilisce il loro funzionamento e di conseguenza regola
alcune attività fondamentali all'interno delle cellule degli organismi viventi. La conoscenza del modo in cui
la catena di amminoacidi che costituisce ciascuna proteina si distribuisce nello spazio,
come i tratti di questa catena si torcano su se stessi
a formare delle strutture elicoidali o si ripieghino a disegnare bracci che si diramano dall'asse centrale,
è cruciale per capire i meccanismi alla base di molte malattie e per progettare farmaci
efficaci. Anche per la pandemia in corso la conoscenza della forma delle proteine che concorrono alle
diverse fasi del ciclo di vita del virus SARS-CoV-2 all'interno della cellula ospite che ha infettato
viene utilizzata per la ricerca e il riposizionamento di antivirali efficaci contro la COVID-19.
Grazie alla cristallografia a raggi X e, più recentemente alla criomicroscopia elettronica,
conosciamo oggi la struttura di circa 170 mila proteine, che però costituiscono solo il 17%
dell'insieme delle proteine presenti nel corpo umano.
Questi metodi sperimentali sono infatti costosi e richiedono tempo.
Per questo motivo da circa cinquanta anni gli scienziati hanno tentato
di risolvere il problema insegnando ai computer a calcolare il modo
in cui gli amminoacidi si dispongono in eliche e bracci partendo dalla
sequenza della proteina, una stringa di lettere di lunghezza variabile
(da qualche decina a qualche centinaio, se si considerano i domini
ovvero le porzioni di proteine che possono essere studiate individualmente).
Ciascuna lettera di questa stringa
può assumere uno tra 21 valori,
codificati in una lettera dell'alfabeto:
A per Alanina, C per la Cisteina, D per l'acido aspartico, e così via fino alla Y di Tirosina.
Hanno cominciato quando il biochimico Christian Anfinsen vinse il Nobel per la chimica nel 1972
dimostrando che le sequenze di amminoacidi determinano
completamente la struttura delle proteine, ma i primi tentativi furono fallimentari.
Calcolare la struttura da principi primi è infatti impossibile. Ciascuna proteina è costituita
da centinaia di amminoacidi e ciascuno di essi, a sua volta, contiene una decina di atomi, e
come questi si dispongano nello spazio dipende dalle interazioni tra di essi stabilite
dalle leggi della fisica. I metodi computazionali si sono quindi orientati verso la ricerca
di somiglianze tra le sequenze di proteine dalla struttura nota per provare a dedurre, statisticamente,
le somiglianze tra le strutture associate. La competizione CASP ha cercato di accelerare il progresso
in questa area creando ed accrescendo nel tempo un database di strutture proteiche note dagli esperimenti
che potessero essere usate come riferimento e stabilendo delle metriche di confronto tra i vari
modelli che via via sono stati sviluppati.
Ogni due anni il comitato scientifico di CASP rende note le sequenze di un campione di circa cento tra proteine
e domini la cui struttura è stata
determinata sperimentalmente ma viene tenuta segreta per tutta la durata della gara.
Nell'arco di alcuni mesi i partecipanti alla competizione calcolano
con i loro modelli le strutture proteiche e le inviano ai giudici del concorso per la valutazione.
Ciascuna struttura viene confrontata con quella sperimentale, la cosiddetta ground truth,
e riceve un punteggio da 0 a 100, chiamato Global Distance Test Total Score (GDT_TS).
Il punteggio viene calcolato partendo dalle percentuali di amminoacidi che,
rispetto alla struttura sperimentale, si trovano a distanze inferiori a 1, 2, 4 e 8 Angstrom (una
lunghezza pari a un decimo di nanometro, cioè alla dimensione di un atomo). Queste quattro percentuali vengono
mediate e così si ottiene il GDT_TS.
Quanto più il GDT_TS è vicino al 100%, tanto più la struttura calcolata è vicina a quella osservata negli esperimenti.
Questo score viene poi
mediato su tutte le strutture del campione per stabilire la performance media del gruppo e,
dopo una procedura di standardizzazione, confrontato con i punteggi ottenuti da tutti gli altri gruppi
per stilare la classifica finale.
|
|
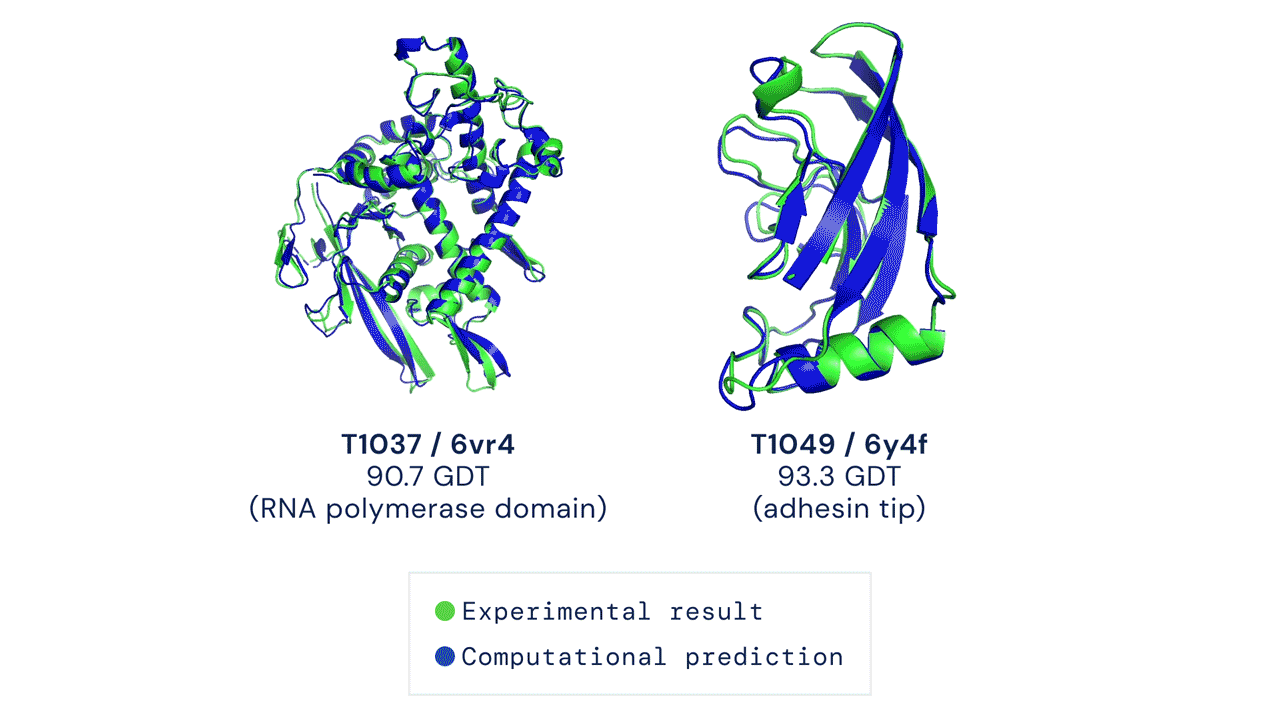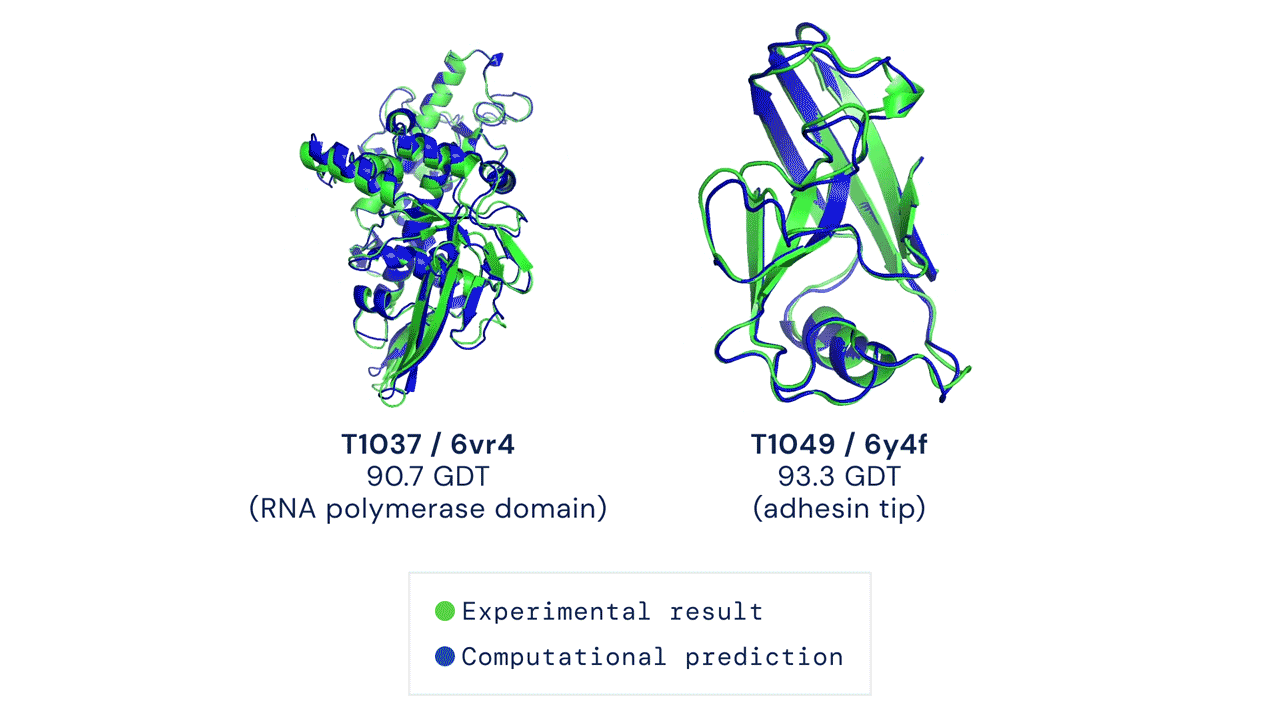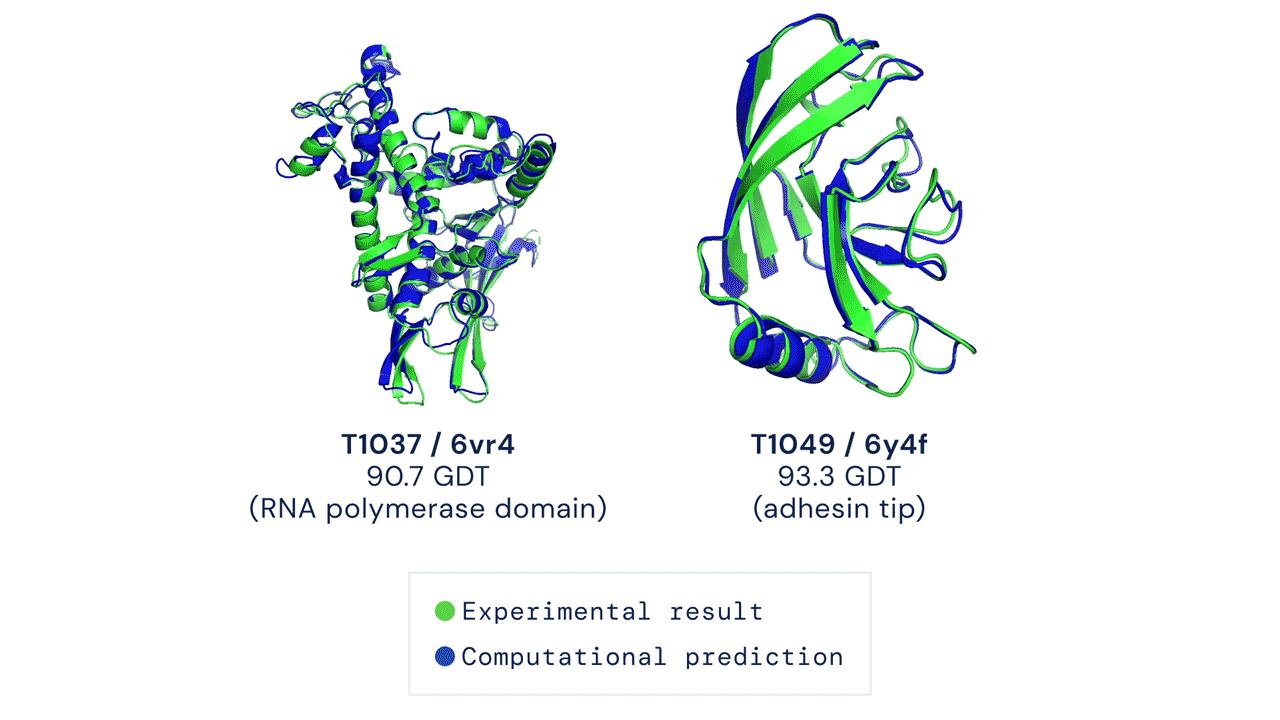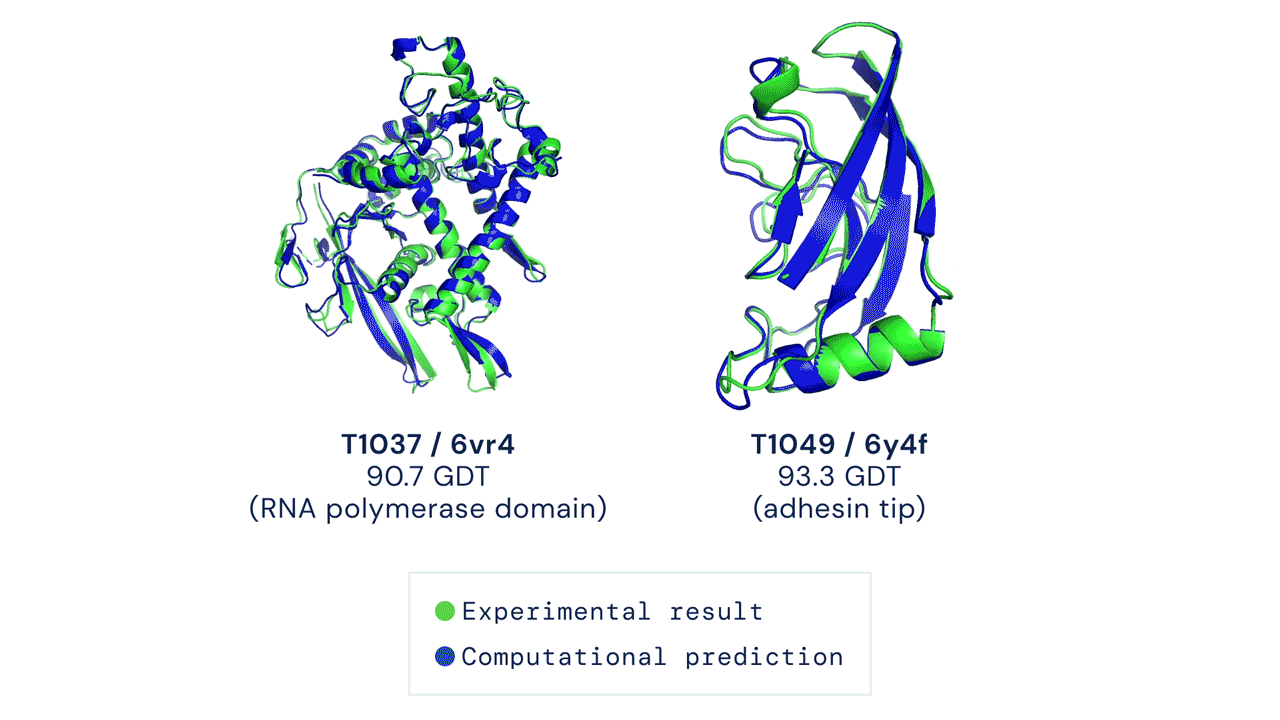 Due esempi di proteine nel campione free modelling della competizione CASP14. Confronto tra la struttura prevista da AlphaFold2 (in blu) e quella sperimentale (in verde). Credit: DeepMind via PhysOrg.
Due esempi di proteine nel campione free modelling della competizione CASP14. Confronto tra la struttura prevista da AlphaFold2 (in blu) e quella sperimentale (in verde). Credit: DeepMind via PhysOrg.
|
Quest'anno l'algoritmo AlphaFold2 ha ottenuto un punteggio medio di 92,4 e per il sottoinsieme di proteine considerate più difficili un punteggio medio di 87, circa 25 punti sopra il secondo gruppo classificato, quello guidato da David Baker della University of Washington. Due terzi delle strutture calcolate da AlphaFold2 hanno ottenuto un punteggio superiore a 90. Questi punteggi possono essere tradotti in termini di RMSD (root mean square deviation) che misura la differenza fra le coordinate atomiche del modello computazionale e quelle della struttura sperimentale. Per AlphaFold2 il valore di RMSD è pari a 1,6 Angstrom. Questo può essere confrontato con quello ottenuto sperimentalmente, che nel caso delle strutture a risoluzione media è di 0,6 Angstrom e può arrivare fino a 0,1-0,2 Angstrom.
"Questi sono gli ordini di grandezza necessari nelle routine di drug discovery, ma visto il salto di accuratezza fatto rispetto al 2018 non sembra improbabile che questi sistemi possano raggiungerli nel prossimo futuro", commenta Silvia Onesti, che dirige la divisione di biologia strutturale del sincrotrone Elettra a Trieste, e aggiunge "il risultato di AlphaFold2 è sorprendente, ma più che vederlo come uno strumento che sostituirà la cristallografia delle proteine, credo possa diventare sinergico. Il lavoro dei biologi strutturali oggi va oltre la ricostruzione della forma delle proteine a partire dai pattern di diffrazione dei raggi X. I ricercatori del mio gruppo dedicano una considerevole quantità di tempo alla produzione e purificazione delle proteine e queste vengono usate solo in parte per gli studi di cristallografia. Un'altra parte viene sfruttata per indagare le loro proprietà biochimiche". La tecnica che per ora sembra meno minacciata dagli avanzamenti di AlphaFold2 è senz'altro quella della microscopia crioelettronica, specializzata nella risoluzione della struttura di grandi complessi proteici. AlphaFold2, infatti, è in grado di calcolare la struttura di proteine che contengono fino ad alcune centinaia di amminoacidi, mentre molti complessi macromolecolari che hanno un ruolo importante in biologia possono contenerne alcune migliaia.
Il risultato di AlphaFold2 rappresenta comunque un enorme passo avanti rispetto alla scorsa edizione di CASP, quella
del 2018, la prima a cui DeepMind ha preso parte con l'algoritmo AlphaFold, realizzando un punteggio medio
poco sotto 60, e posizionandosi circa 6 punti sopra il secondo classificato.
"Pensavo avremmo dovuto aspettare dieci anni per arrivare
dai risultati di AlphaFold del 2018 a quelli di quest'anno.
Siamo vicini al limite fisico dell'accuratezza.
Fondamentalmente queste strutture sono flessibili. Non ha senso parlare di risoluzioni superiori",
ha dichiarato a MIT Technology Review
Mohammed AlQuraish, biologo computazionale della Columbia University e uno dei partecipanti alla competizione.
AlphaFold2 ha stracciato tutti gli avversarsi anche secondo un'altra metrica, che considera distanze più piccole
rispetto al punteggio che abbiamo descritto prima e serve a valutare l'accuratezza della struttura in maniera ancora
più stringente. Andrei Lupas, biologo evoluzionista del Max Planck Institute for Developmental Biology a Tubinga in Germania,
ha guidato la valutazione 'High Accuracy', presentando i risultati martedì 1 dicembre.
Tuttavia, entrambe queste metriche misurano l'accuratezza globale della struttura e non quella locale, come spiega AlQuraish in un editoriale del 2019 sulla rivista Bioinformatics, commentando i risultati
di AlphaFold nella scorsa edizione di CASP: "l'accuratezza a livello locale relativa, per esempio
al coordinamento nel movimento degli atomi in un sito attivo o al cambiamento localizzato
di conformazione dovuta a una mutazione, può essere l'aspetto più importante di una struttura
per rispondere a un insieme più ampio di domande biologiche".
I risultati di AlphaFold2 restano sorprendenti, quasi incredibili,
così
Lupas ha voluto testarli ulteriormente, sottoponendogli la
sequenza di una proteina studiata nel suo laboratorio che, nonostante la disponibilità dei dati di diffrazione dai raggi X, non era riuscito a risolvere.
Grazie alla struttura calcolata da AlphaFold2, i
ricercatori del gruppo di Lupas hanno ottenuto un modello computazionale della loro proteina che gli ha permesso
di fasare e interpretare i dati di diffrazione: "È praticamente perfetta, non ho idea di come ci riescano",
ha detto a Science.
Per ora nessuno sa come DeepMind sia riuscito a ottenere questi risultati sorprendenti.
Demis Hassabis, CEO della società, ha dichiarato che i dettagli sul funzionamento di AlphaFold2 saranno resi
noti in un articolo scientifico che probabilmente verrà pubblicato il prossimo anno.
Martedì, secondo quanto riportato sul programma della conferenza,
John Jumper, team leader di AlphaFold2, ha presentato i risultati della sua squadra e potrebbe aver dato qualche dettaglio in più,
ma finora le sue slides non sono state archiviate insieme a quelle degli altri gruppi.
Qualche informazione ci arriva dalle dichiarazioni di Jumper e dall'abstract pubblicato qui.
Il cuore dell'algoritmo è una rete neurale profonda con un'architettura nuova, chiamata attention based,
capace di concentrarsi su porzioni più piccole della struttura proteica e poi assemblare ciò
che ha appreso per prevedere la forma completa, come si farebbe con i pezzi di un puzzle.
Tuttavia, pare che le differenze fra AlphaFold2 e AlphaFold siano limitate alla componente di deep learning
dell'algoritmo, che è solo una parte dell'intero sistema. Possiamo, dunque, riferirci ad AlphaFold
per parlare dell'impostazione generale del problema.
La struttura della proteina viene schematizzata come una catena di amminoacidi e la
sua geometria viene descritta
dalle distanze tra ciascuna coppia di amminoacidi (2L variabili se L è la lunghezza della sequenza) e una coppia
di angoli di torsione per ciascun amminoacido (4L variabili in tutto).
AlphaFold è composto di tre passi. Nel primo si estraggono le caratteristiche delle sequenze di amminoacidi
che sono rilevanti per la struttura finale assunta dalla proteina. Questo si fa cercando nel database su cui si
svolge l'apprendimento, costituito dalle 170 mila proteine contenute nel Protein Data Bank, delle altre sequenze che
hanno porzioni simili. Si costruisce così un insieme di omologhi della proteina che si vuole studiare,
insieme che condivide un antenato comune. Ricostruendo il processo di evoluzione, si è in grado di
codificare le informazioni rilevanti per la struttura della proteina che sono contenute nella sua sequenza genetica.
Nel secondo passo queste informazioni vengono date in pasto a una rete neurale profonda che le utilizza per prevedere
le distribuzioni probabilistiche delle distanze tra tutte le coppie di amminoacidi presenti nella sequenza
e dei loro angoli di torsione. Le distribuzioni probabilistiche sono sostanzialmente degli istogrammi che dicono
qual è la probabilità che la distanza tra due specifici amminoacidi sia contenuta in un certo intervallo di valori.
Queste distribuzioni vengono utilizzate dal terzo passo dell'algoritmo, che calcola la posizione di ciascun amminoacido
che meglio rispetti i vincoli stabiliti dalle distribuzioni su distanze e angoli. Questo calcolo è effettuato con metodi numerici
convenzionali. La struttura teorica così ottenuta viene confrontata con quella sperimentale e, mediante un processo iterativo,
i pesi della rete neurale vengono aggiustati per ottenere la migliore corrispondenza possibile tra le due.
Per AlphaFold era stata utilizzata una rete neurale con una architettura molto sofisticata, chiamata
dilated residual convolutional neural network, descritta in dettaglio in questo articolo
pubblicato su Nature all'inizio del 2020. Si stratta di una rete neurale molto profonda,
con 220 strati interni e circa 21 milioni di parametri che descrivono le connessioni tra i nodi
disposti sui diversi strati.
È importante sottolineare come il lavoro di DeepMind, prima con AlphaFold e poi con AlphaFold2,
sia stato costruito su due decenni di progressi accumulati nel campo della biologica computazionale.
Da una parte il metodo basato sulla ricerca degli omologhi e dei meccanismi di co-evoluzione,
dall'altra i software che traducono le informazioni su distanze e angoli di torsione in strutture tridimensionali.
DeepMind ha però portato questi strumenti su un'altra scala, raggiungendo risultati che solo due anni fa
sarebbero stati impensabili. Inoltre, ha influenzato l'intero campo di ricerca: quest'anno circa la metà dei gruppi
partecipanti ha impiegato qualche forma di deep learning nei suoi algoritmi.
Dame Janet Thornton, direttrice emerita dello European Bioinformatics Institute non coinvolta nella competizione
CASP, ha dichiarato: "Cominciavo a pensare che non avrei visto la soluzione al problema
nell'arco della mia vita. Una maggiore comprensione della struttura delle proteine e la
possibilità di prevederle usando un computer, aprirà la strada a nuove scoperte nel campo dell'evoluzione e della medicina,
come per esempio comprendere come variazioni genetiche osservate tra le persone siano legate all'insorgenza di alcune malattie".
Perché questo scenario si realizzi è necessario che AlphaFold2 venga condiviso con la comunità scientifica
e che sia utilizzabile nella pratica dei laboratori. Demis Hassabis, il CEO di DeepMind, ha dichiarato che
intende rendere AlphaFold2 accessibile a tutti i ricercatori. Per ora il training dell'algoritmo richiede alcune
settimane usando il potere computazionale equivalente a quello di 100 o 200 GPU e impiega alcuni giorni per determinare
la struttura di una singola proteina (ricordiamo che i complessi di proteine sono per ora fuori dalla sua portata).
Di certo AlphaFold2 segna un cambio di passo per DeepMind. La società londinese finora
si era concentrata sulla soluzione di problemi matematicamente molto complessi,
come il gioco degli scacchi o del Go, ma il suo obiettivo di lungo periodo
è quello di sviluppare la cosiddetta artificial general intelligence.
Non sappiamo se il risultato appena ottenuto sia un passo importante
in quella direzione, ma di certo promette di avere un impatto enorme sul modo di fare ricerca
nel campo della biologia e della medicina.
|
|
|
|
|
Per suggerimenti e commenti scrivere a: [email protected]
|
|
Per donare alla Newsletter clicca qui
|
|
Se invece non vuoi più ricevere la newsletter clicca qui
|
|
PS: per tenere Scienza in rete fuori dallo spam aggiungi la nostra mail [email protected]
tuoi contatti (per Gmail, vai a contacts.google.com e clicca su "crea contatto").
Se Scienza in rete finisce nelle promozioni di Gmail, trascinala nella casella della tua posta
in entrata per non perdere mai un numero!
|
|
Segui Scienza in rete
|




|
|
|
By:
|

|
|
|


